Corona-IgG-Blot - Screening auf endemische Coronaviren und SARS-CoV-2
Einführung
Coronaviren sind unter Säugetieren und Vögeln weit verbreitet. Bei den sieben bekannten humanpathogenen Coronavirus-Spezies (HCoV) werden die zwei Genera unterschieden:
- Alphacoronaviren
• HCoV-229E
• HCoV-NL63 - Betacoronaviren
• HCoV-HKU1
• HCoV-OC43
• SARS-CoV
• MERS-CoV
• SARS-CoV-2
Vier dieser Spezies zirkulieren weltweit endemisch: HCoV229E, HCoV-NL63, HCoV-HKU1 und HCoV-OC43. Sie verursachen vorwiegend milde Erkältungskrankheiten, können aber mitunter schwere Pneumonien hervorrufen, vor allem im frühen Kindesalter sowie bei alten und immunsupprimierten Menschen. SARS-CoV-2 (Severe acute respiratory syndrome coronavirus type 2) ist ein neues Coronavirus, das Anfang 2020 als Auslöser der COVID-19-Erkrankung identifiziert wurde.
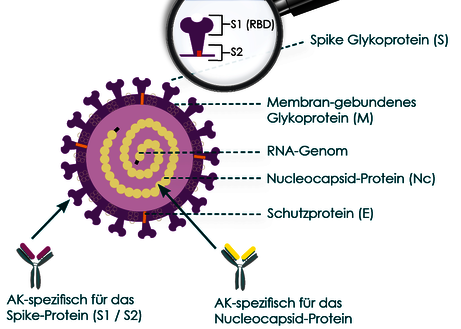
Coronaviren sind RNA-Viren. Die an N-Proteine (Nukleoproteine) gekoppelte RNA bildet das Nucleokapsid (Nc), dieses ist von einer Membran umhüllt, die mit Spikeproteinen bestückt ist.
Das Spikeprotein ist für den Eintritt in die Wirtszelle zuständig und besteht aus zwei Untereinheiten: Die S1-Untereinheit enthält die Receptor binding domain (RBD), die an den Wirtszellrezeptor bindet; die S2-Untereinheit vermittelt danach die Fusion von Virushülle und Zellmembran (Abb. 1).
Die Spike- und Nukleokapsid-Proteine von SARS-CoV-2 sind hochimmunogen. Antikörper gegen die Rezeptorbindungsdomäne (RBD) des Spike-Proteins sind neutralisierend (protektiv). Aus diesem Grund ist S1 Grundlage der Vektor- und RNA-Impfstoffe, IgG-Antikörper gegen S2 und Nc werden in diesem Fall nicht gebildet. Im Gegenzug dazu kommt es bei einer Infektion jedoch häufig zur Bildung spezifischer IgG-Antikörper gegen Nc.
| Bezeichnung | Quelle |
|---|---|
| SARS-CoV-2-S1 | Rekombinantes SARS-CoV-2-SpikeProtein Domäne 1 |
| SARS-CoV-2-S2 | Rekombinantes SARS-CoV-2-SpikeProtein Domäne 2 |
| SARS-CoV-2-Nc | Rekombinantes SARS-CoV-2-Nukleokapsid-Protein |
| HCoV-HKU1-Nc | Rekombinantes HCoV-HKU1-Nukleokapsid-Protein |
| HCoV-OC43-Nc | Rekombinantes HCoV-OC43-Nukleokapsid-Protein |
| HCoV-NL63-Nc | Rekombinantes HCoV-NL63-Nukleokapsid-Protein |
| HCoV-229E-Nc | Rekombinantes HCoV-229E-Nukleokapsid-Protein |
Tab. 1 Untersuchte Antigene im Corona-IgG-Blot
Methode
Der Corona-IgG-Immunoblot (EUROLINE, Fa. Euroimmun) ermöglicht die simultane Bestimmung von IgG gegen die SARS-Cov-2-Antigene Spike-Protein (S1 und S2) und Nucleocapid-Protein (Nc) sowie in demselben Untersuchungsgang von IgG gegen die endemischen Coronaviren HCoV-229E, HCoV-NL63, HCoV-HKU1 und HCoV-OC43 (Tab. 1).
Klinische Indikation
Es wurde gezeigt, dass Menschen auch ohne durchgemachte SARS-CoV-2-Infektion eine kreuzreaktive T-Zell-Antwort gegen dieses Virus aufweisen können. Diese Immunantwort wird wahrscheinlich durch vorherige Infektionen mit den saisonalen Coronaviren (OC43, 229E, HKU1 und NL63) hervorgerufen.
So zeigen 2 aktuelle Studien einen direkten Zusammenhang zwischen früheren Infektionen mit den saisonalen Coronaviren und dem Schweregrad einer COVID-19-Erkrankung. Hierfür wurden Serumproben von Patienten mit COVID-19 auf das Vorliegen von Antikörpern gegen die saisonalen Coronaviren untersucht. Patienten mit einer schweren COVID-19-Erkrankung wiesen eine sehr viel schwächere Antikörperantwort gegen die endemischen Coronaviren auf. Besonders ausgeprägt war dieser Zusammenhang bei der Antwort gegen das Coronavirus OC43. Eine fehlende Immunantwort gegen saisonale Coronaviren könnte somit einen zusätzlichen Risikofaktor für einen schweren COVID-19-Verlauf darstellen (Abb. 2).
Abb. 2 Musterbefund Corona-IgG-Blot zum Screening von IgG-Antikörpern gegen endemische Coronaviren und SARS-CoV-2. In Studien zeigten Patienten mit geringen bzw. fehlenden IgG-Antikörpern gegen HCoV (ins besondere gegen OC43) ein höheres Risiko für eine schwere Covid-19 Erkrankung als Patienten bei denen diese Antikörper nachgewiesen wurden.
Die Bestimmung von Antikörpern gegen die SARS-CoV-2-Antigene S1, S2 und Nc ermöglicht die Bestätigung von SARSCoV-2-Infektionen nach Rekonvaleszenz (3 bis 4 Wochen nach Infektion). Allerdings ist die quantitative Bestimmung von IgG gegen S1 (Quantivac, Fa. Euroimmun) die sensitivere Nachweismethode und insbesondere für Verlaufsmessungen besser geeignet.
Material
1 ml Serum
Der Transport ins Labor ist nicht zeitkritisch und kann per Postversand erfolgen.
Abrechnung
Eine Abrechnung ist nur im privatärztlichen Bereich (GOÄ) gegeben. Die Test-Kosten für Selbstzahler entnehmen Sie bitte dem PDF-Dokument.
Literatur
- Udugama B, Kadhiresan P, Kozlowski HN et al. Diagnosing COVID-19: The Disease and Tools for Detection. ACS Nano. 2020 Apr 9.
- Cheng MP, Papenburg J, Desjardins et al. Diagnostic Testing for Severe Acute Respiratory Syndrome-Related Coronavirus-2: A Narrative Review. Ann Intern Med. 2020; 172(11): 726-34
- Cheng MP, Yansouni CP, Basta NE et al. Serodiagnostics for Severe Acute Respiratory Syndrome-Related Coronavirus 2: A Narrative Review. Ann Intern Med. 2020; 173(6): 450-60
- Dugas M, Grote-Westrick T, Merle U et al. Lack of antibodies against seasonal coronavirus OC43 nucleocapsid protein identifies patients at risk of critical COVID-19. J Clin Virology 2021; 104847
- Dugas M, Grote-Westrick T, Vollenberg R et al. Less severe course of COVID19 is associated with elevated levels of antibodies against seasonal human coronaviruses OC43 and HKU1. Int J Infectious Diseases 2021; 105: 304-306;