Genetik und immunologische Funktionen des HLA-Systems
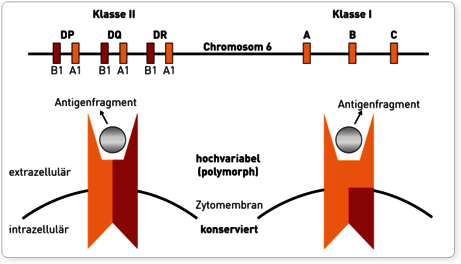
Die HLA-Oberflächenmoleküle werden durch eng gekoppelte Gene auf dem menschlichen Chromosom 6 kodiert. Dieser Abschnitt wird auch als Haupthistokompatibilitätskomplex (engl.: major histocompatibility complex, MHC) bezeichnet, da er im Rahmen der Transplantationsforschung entdeckt wurde. Transplantiert man Gewebe von einem Individuum auf ein genetisch differentes Individuum derselben Spezies, wird das Transplantat als fremd erkannt und ohne immunsuppressive Maßnahmen abgestoßen. Das Ausmaß der immunologischen Abwehrreaktion hängt wesentlich davon ab, inwieweit sich Spender und Empfänger in ihren auf dem MHC kodierten Transplantationsantigenen unterscheiden. Bei allen Vertebraten wurde ein MHC-System nachgewiesen, beim Menschen wird dieser spezifische Genabschnitt als HLA bezeichnet. Der gesamte HLA-Genkomplex umfasst ca. 4.000 Kilobasen (kb) und kann in 2 „klassische“ Gen-Regionen unterteilt werden: Die Region der Klasse I-Gene kodiert für die HLA-Merkmale A, B und C. Zu den Klasse II-Genen gehören die HLA-Merkmale DR, DQ und DP.
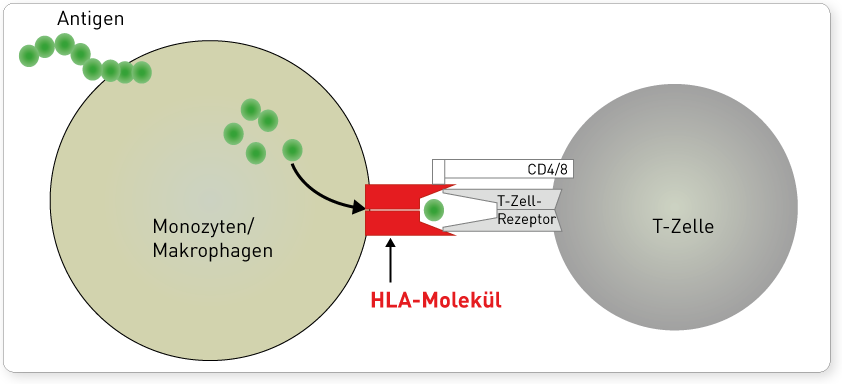
Die Einteilung in zwei Klassen basiert auf funktionellen Unterschieden im Bereich der Antigenpräsentation. HLA-Moleküle werden in der Zelle mit Antigenfragmenten beladen und präsentieren diese an der Zelloberfläche. Zytotoxische CD8-T-Lymphozyten erkennen nur an HLA-Klasse I-Moleküle gebundene Antigene, wodurch die klonale Vermehrung dieser T-Lymphozyten initiiert wird (zelluläre Immunantwort). CD4-T-Lymphozyten dagegen interagieren mit Antigenen, die auf HLA-Klasse-II-Molekülen präsentiert werden. Sie regen nach ihrer Aktivierung unter anderem B-Lymphozyten zur Ausdifferenzierung in Plasmazellen an (humorale Immunantwort). Somit werden auf HLA-Klasse-I vorrangig intrazellulär synthetisierte Proteinefragmente (Viren, intrazelluläre Bakterien, körpereigene Peptide) präsentiert, während auf HLA-Klasse-II vor allem phagozytierte extrazelluläre Proteine (z.B. extrazelluläre Bakterien) gebunden werden. Diese funktionelle Unterscheidung erklärt auch, dass HLA-Klasse-I-Moleküle auf fast allen kernhaltigen Zellen und Thrombozyten exprimiert werden, denn potentiell kann jede Körperzelle von Erregern befallen und ihre Zerstörung notwendig werden. Die Expression von HLA-Klasse-II-Molekülen hingegen ist auf phagozytierende Zelltypen wie Monozyten und Makrophagen sowie B-Lymphozyten beschränkt.
Jeder Mensch trägt ein individuelles HLA-Muster
Das HLA-System ist hochpolymorph, d.h. für die meisten HLA-Moleküle existieren zahlreiche Varianten, sogenannte Allele. Da jeder Mensch jeweils ein Chromosom von der Mutter und eines vom Vater ererbt, besitzt er für jedes HLA-Merkmal jeweils 2 Allele (z.B. B27 und B15). Die Vielfalt der HLA-Moleküle und ihrer Kombinationen untereinander sorgen für ein nahezu individuelles HLA-Muster jedes Menschen. Durch molekulargenetische Analysen ist bekannt, welche Bereiche der exprimierten HLA-Moleküle hochvariabel sind und wo diese Abschnitte des Proteins kodiert werden. Dieses ist für molekulargenetische HLA-Typisierungen entscheidend, da konservierte Bereiche dafür ungeeignet sind. Bei HLA-Klasse-I-Molekülen wird im MHC-Komplex nur die polymorphe α-Kette kodiert. Diese α-Kette verbindet sich mit der auf Chromosom 15 kodierten konservierten β-Kette zum aktiven Protein. Die HLA-Klasse-II-Region auf Chromosom 6 ist hingegen durch eine ausgeprägte Polygenie gekennzeichnet. Für die HLA-DR-Moleküle gibt es ein nicht polymorphes α-Kettengen (DRA) und mehrere ß-Kettengene (DRB1-DRB9), wobei nur das DRB1-Gen zur Grundausstattung eines jeden Menschen gehört. Die Anzahl der restlichen DRB-Gene schwankt je nach Haplotyp. In der HLA-DQ-Region liegen zwei α- und zwei β-Kettengene vor, von denen DQA1 und DQB1 hochgradig polymorph sind, während die anderen beiden Gene DQA2 und DQB2 nur Pseudogene sind. In der DP-Region werden die exprimierten DP-Moleküle DPA1 und DPB1 kodiert, zusätzlich liegen die Pseudogene DPA2, DPA3 und DPB2 vor.
Nomenklatur des HLA-Systems
Da bei molekulargenetischen Typisierungen immer nur die polymorphen Genabschnitte, d.h. die Abschnitte, in denen sich die Individuen jeweils unterscheiden, untersucht werden, ergibt sich folgendes international vereinbartes Vorgehen: bei HLA-A, -B und -C wird immer nur das α-Ketten-Gen untersucht, bei HLA-DR das β1-Kettengen (DRB1) und bei HLA-DQ die DQA1- und DQB1-Gene. Dieses Vorgehen spiegelt sich auch in der Nomenklatur wieder. Zum jetzigen Zeitpunkt sind mehr als 6800 HLA-Allele weltweit bekannt, die sich u.a. zusammensetzen aus 1698 A-, 2271 B-, 1213 C-, 975 DRB1, 44 DQA1, 158 DQB1, 32 DPA1 und 149 DPB1-Allelen. Grundsätzlich sind einzelne Allele in einer Population nicht gleich verteilt. Es gibt häufigere und seltenere Allele. Die Frequenz der Allele kann in ethnisch verschiedenen Bevölkerungsgruppen extrem variieren:
Die Analyse der HLA-Antigene erfolgt aufgrund des historischen Ursprungs „zweigleisig“. Zunächst wurden HLA-Merkmale serologisch mittels spezifischer Antikörper bestimmt. Diese Methode weist direkt auf der Zelloberfläche exprimierte HLA-Moleküle auf Proteinebene nach, ist aber durch den Mangel an hochspezifischen Antiseren bzw. HLA-Antikörpern limitiert. Die Einführung molekularbiologischer Nachweismethoden auf DNA-Ebene seit Mitte der 80er Jahre hat die Unterscheidung der Vielzahl von Allelen entscheidend verbessert. Selbst geringfügige Unterschiede in den DNA-Sequenzen von HLA-Allelen sind immunologisch relevant, können aber aufgrund der begrenzten Epitopspezifität von Antikörpern mit serologischen Methoden auf Proteinebene nicht erfasst werden. So wurden z.B. für das serologische HLA-Merkmal DR4 inzwischen durch molekularbiologische Verfahren mindestens 102 Subtypen nachgewiesen. Damit wurde eine neue Nomenklatur erforderlich. So bedeutet z.B. HLA-DRB1*04:02, dass der HLA-Lokus DR und dort der polymorphe β-Ketten-Bereich (B1) untersucht wurde. Danach wird, getrennt durch einen Stern, zunächst die Hauptgruppe 04 (d.h. serologisch DR4) angegeben, gefolgt vom Subtyp 02. Eine sechsstellige Angabe erfolgt, wenn ein Allel nachgewiesen wird, welches mit dem genannten identisch ist und sich nur durch eine stumme Mutation, die keine Änderung in der Aminosäuresequenz zur Folge hat, unterscheidet (z.B. DRB1*04:02:01).
molekularbiologisch |
| serologisch |
|---|---|---|
HLA-DR | Genlokus | HLA-DR |
HLA-DRB1 | Spezielle Region | |
HLA-DRB1*13 | Hauptgruppe | HLA-DR13 |
HLA-DRB1*13:02 | Subgruppe |
Molekularbiologische und serologische Nomenklatur des HLA-Merkmals DR13
Literatur
- Marsh SGE, Parham P and Barber LD. The HLA FactsBook. Academic Press. 2003, San Diego.
- Begovich AB et al. Polymorphism, recombination, and linkage disequilibrium within the HLA class II region. J Immunol. 1992; 148:249 – 258.
- Klein J, Sato A. The HLA system. First of two parts. N Engl J Med. 2000; 343(10):702-9.
- Robinson J, et al. The IMGT/HLA database. Nucleic Acids Res. 2011; 39: D1171-6.
- Albert E. Immungenetik. In: Gemsa, D, Kalden JR, Resch K, Hrsg. Immunologie. Grundlagen – Klinik - Praxis. Stuttgart: Thieme; 1997. S. 87-120.